La terminazione della duplicazione del DNA nei batteri è stata studiata su Escherichia coli. Il DNA di E. coli contiene alcune sequenze definite ter. Queste zone legano delle proteine tus (ter utiliziation substances) che legandosi al filamento di DNA bloccano l'attività del sistema di duplicazione.
DNA ligasi
È possibile ipotizzare che nel filamento veloce la polimerasi possa duplicare il DNA senza mai staccarsi; nel filamento lento a causa della formazione dei frammenti di Okazaki questo non avviene. In altre parole, il filamento lento è formato da una serie di filamenti di DNA interrotti (da ricordare che i filamenti di start di RNA sono stati rimossi da una DNA polimerasi I). Questi segmenti sono interrotti e devono essere riuniti; per compiere questo lavoro interviene una DNA ligasi che forma i classici ponti fosforici 5'→3'.
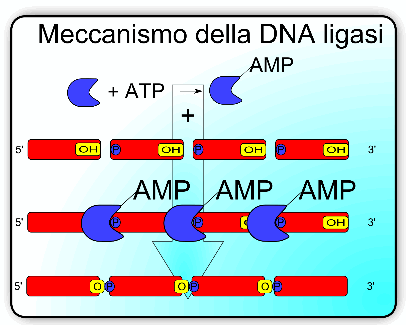
Meccanismo di azione e funzionamento delle DNA ligasi.
Ripristino dei telomeri
La maggior parte dei batteri possiedono il codice genetico organizzato in modo circolare. In questo modo è ipotizzabile che successivamente alle fasi della duplicazione i filamenti neosintetizzati possano unirsi semplicemente mediante la ligasi che, facilitata dalla vicinanza delle due estremità, potrebbe interagire con il DNA e circolarizzarlo. Negli eucarioti il DNA è perlopiù lineare e le estremità 5'→3' si troverebbero perse subito dopo l'intervento di rimozione del frammento di RNA ad opera della DNA polimerasi I. Questa situazione è inammissibile in quanto a seguito di ripetute divisioni cellulari le estremità si accorcerebbero sempre di più con conseguenze disastrose per la cellula. Il ripristino delle sequenze altrimenti perse viene affidato a enzimi di natura riboproteica chiamati telomerasi.
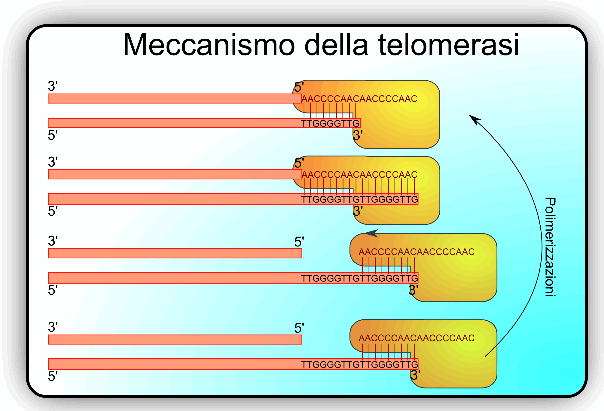
Meccanismo di funzionamento delle telomerasi.
Introduzione all'informazione genetica:
DNA e
RNA,
cromosoma,
cromatina,
genoma.
Trascrizione:
RNA polimerasi batterica,
allungamento dell'RNA,
Trascrizione eucariotica,
terminazione della trascrizione,
RNA interference (
pre-mRNA,
siRNA).
Traduzione:
Ribosoma,
RNA ribosomiale,
ribozima,
trascrittoma.
Duplicazione del DNA:
Primosoma,
forcella di replicazione,
DNA polimerasi I procariotica,
DNA polimerasi III procariotica,
Terminazione della duplicazione del DNA.
Tecniche e laboratorio PCR,
LCR,
Real time PCR.
Feedback
Il tuo aiuto è importante. Ti chiediamo un minuto per rispondere a questo breve sondaggio
Come valuteresti questo articolo?
Vuoi suggerirci qualcosa?