La traduzione comprende tutti gli eventi, di natura enzimatica, che portano all'effettiva espressione di una proteina. L'RNA prodotto nelle fasi della trascrizione è elaborato da complessi organuli cellulari chiamati ribosomi costituiti in larga parte da proteine e da RNA ribosomiale.
La traduzione deve essere un processo veloce ed efficiente poiché piccole modifiche della struttura primaria del polipeptide possono portare alla formazione di proteine non funzionali con conseguenze più o meno gravi per la cellula e per l'organismo intero.
I processi biochimici che rendono possibile la traduzione sono molto differenti tra i procarioti e gli eucarioti. La traduzione negli eucarioti è molto più complessa e coinvolge molti più elementi rispetto a quella che si osserva nei procarioti.
La traduzione è, convenzionalmente, divisa in tre fasi: inizio, allungamento e terminazione. Esistono delle differenze nella traduzione dei procarioti e degli eucarioti sia dal punto di vista del metodo enzimologico utilizzato che dal punto di vista della localizzazione intracellulare. La traduzione procariotica è più semplice e coinvolge meno enzimi e viene operata nel liquido intracellulare. Negli eucarioti il pool enzimatico è più complesso e i processi di sintesi proteica, che è un sinonimo di traduzione, avvengono nel citoplasma; per questo motivo il trascritto di mRNA deve essere dapprima traslocato dal nucleo al citoplasma e successivamente tradotto.
Traduzione nei procarioti
La molecola di mRNA viene tradotta nei ribosomi procariotici che consistono di due unità definite 30S e 50S, i ribosomi batterici sono più piccoli rispetto ai ribosomi eucariotici di circa il 10% e sono costituiti da RNA ribosomiale ed una aliquota di proteine.
L'inizio della traduzione è particolare. Il primo aminoacido della catena polipeptidica è una metionina che è codificata, a livello dell'mRNA, dal codone AUG. Tuttavia l'inizio della traduzione nei procarioti è particolare in quanto la prima metionina ha la caratteristica di essere formilata a livello dell'azoto.
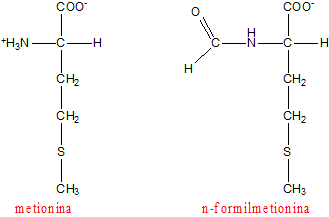
Metionina ed n-formil metionina
La n-formilmetionina è prodotta a partire dalla metionina che viene processata grazie all'opera di un enzima chiamato metionina-formilasi.
Il passo successivo è il caricamento del tRNA che rappresenta quel particolare RNA adibito al trasporto dell'aminoacido fino al ribosoma. Le molecole di tRNA vengono “caricate” del proprio aminoacido specifico in una reazione enzimatica che consuma una molecola di ATP.
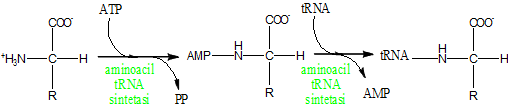
Caricamento del tRNA con un aminoacido generico.
È importante ricordare che ogni tRNA è specifico per un singolo aminoacido, ragion per cui è estremamente necessario che l'appaiamento aminoacido-aminoacido-tRNA sia specifico. Se così non fosse in fase di traduzione verrebbe inserito un aminoacido al posto sbagliato con il rischio di rendere la proteina meno funzionale o, in alcuni casi, totalmente non funzionale.
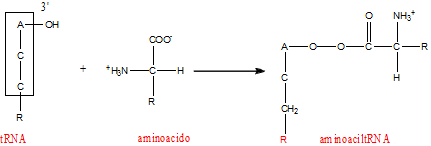
Aggancio del tRNA.
L'ossidrile libero in 3' del braccio accettore del tRNA è il punto nel quale avviene l'aggancio dell'aminoacido.
Fattori d'inizio dei procarioti
Per poter operare la sintesi proteica i ribosomi hanno bisogno di alcune molecole definite fattori iniziali di traduzione.
Queste fattori vengono chiamati con gli acronimi IF1, IF2 ed IF3, dove “I” sta per initial ed “F” per factor. Il loro ruolo è fondamentale poiché svolgono alcuni compiti di aiuto nella preparazione del ribosoma e nel legame della opportuna subunità ribosomica alla catena di RNA.
Fattori di trascrizione dei procarioti
| Fattore |
Ruolo |
|---|
| IF1 |
Allontana le subunità 30S e 50S del ribosoma. |
| IF2 |
Promuove il legame del complesso metionina formilata-tRNA al ribosoma 30S. Richiede GTP che, tuttavia, sembra non essere idrolizzato. |
| IF3 |
Lega l'mRNA alla subunità 30S e, al contempo, impedisce il riaggancio con la subunità 50S. |
Ruolo dei fattori iniziali per l'inizio della traduzione nei procarioti
Il primo passo per poter operare la traduzione è rappresentato dalla separazione del ribosoma nelle sue due unità costituenti 30S e 50S. La separazione del ribosoma è coadiuvata da IF1. In questo modo la subunità libera 30S è capace di legare a sé, grazie all'ausilio di IF3, la catena di mRNA. Un punto triviale riguardante la formazione di questo legame è dato dalla regione di aggancio: esiste una sequenza specifica nell'mRNA da tradurre oppure il ribosoma è capace di legare l'mRNA in qualsiasi punto contenente una tripletta AUG? La risposta è che esiste una specifica sequenza definita sequenza di Shine-Dalgarno nella quale la subunità 30S del ribosoma si lega. La sequenza di Shine-Dalgarno è la seguente:
AGGAGG
Questa sequenza si lega, a livello dell'RNA ribosomiale 16 S con la sua antisequenza che è:
UCCUCC
In questo modo il codone di start AUG, che è molto vicino alla sequenza consensus appena vista, può essere letto.
Traduzione negli eucarioti
L'inizio della traduzione eucariotica è molto più complesso. Dopo la trascrizione, l'mRNA è processato mediante gli eventi di “maturazione” che consistono nel capping e nella poliadenilazione. Questi eventi servono per aumentare il livello di traduzione e sono assenti nei procarioti.
Esistono differenze sostanziali tra i fattori di inizio della traduzione rispetto ai procarioti. Negli eucarioti ci sono un numero maggiore di elementi che coadiuvano l'inizio del processo, questi elementi rendono più complesso tutto il sistema della traduzione. C'è da considerare che l'mRNA eucariotico presenta alcune strutture peculiari tra cui il CAP ed una potenziale forcina che devono essere trattate affinché il ribosoma possa compiere il proprio lavoro.
|
Fattore
|
Ruolo
|
|
EIF4F
|
Riconosce il CAP e, grazie ad una attività elicasica, svolge la forcina dell'mRNA.
|
|
EIF2
|
Coadiuva il legame dell'mRNA alla particella 40S del ribosoma. Aggancia a sé un GTP.
|
|
EIF2B
|
Serve per l'idrolosi del GTP in GDP presente in EIF2; in questo modo il fattore EIF2 si rilascia dal complesso ribosomico.
|
|
EIF3
|
Si lega alla subunità 40S del ribosoma e ne impedisce l'aggancio con la subunità 60S.
|
|
EIF6
|
Si lega alla subunità 60S del ribosoma e ne impedisce l'aggancio con la subunità 40S.
|
|
EIF5
|
Coadiuva l'aggancio della subunità 40S e della subunità 60S del ribosoma.
|
|
EIF1, EIF1A
|
Coadiuvano il “movimento” del complesso ribosomico lungo la catena di mRNA.
|
Fattori di inizio eucariotici
Se nei procarioti il primo aminoacido incorporato nella neonascente catena polipeptidica è la n-formilmetionina negli eucarioti il corrispettivo aminoacido è la metionina. Il codone principale di start è sempre AUG ma c'è una forma diversa che riguarda il modo con il quale il ribosoma arriva a questo codone rispetto a quanto avviene nei batteri. Esperimenti hanno dimostrato che esiste una sequenza consensus altamente conservata che circonda il codone di start. La sequenza prende il nome dalla sua scopritrice Marylind Kozak che la propose nel 1975. Questa sequenza è:
CCA/GCCAUGC
È da notare che il codone AUG codificante per la metionina è espresso nella sequenza di Kozak.
Esistono delle variazioni a questa sequenza che, comunque, è un modello che fa parte delle cosiddette regole di Kozak che determinano la formazione ideale di un ORF, acronimo di un open reading frame. Un ORF non è altro che una parte dell'mRNA che, potenzialmente, può codificare per una proteina; questa sequenza è data dalla presenza di una zona di “inizio” e da un terminatore, la cui struttura verrò proposta in avanti.
Allungamento
Nella fase di allungamento alla neonascente catena polipeptidica vengono aggiunti gli aminoacidi la cui informazione, in termini di sequenza, è conservata nell'mRNA.
È opportuno ricordare che il codice genetico è di tipo non sovrapponibile perché i nucleotidi non sono correlati a più di un codone, e non interrotto per cui la sequenza di mRNA viene tradotta senza ambiguità ed interruzioni a meno di errori nell'apparato di trascrizione.
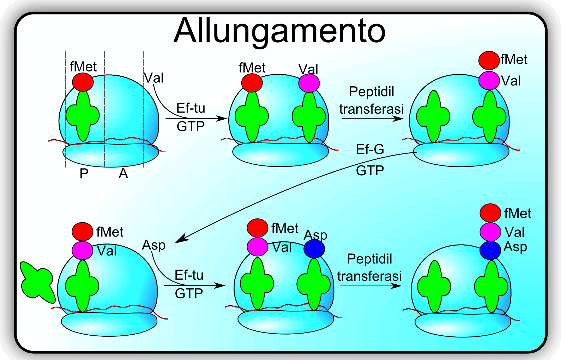
Allungamento della catena polipeptidica
Il ribosoma è il luogo dove viene operato l'allungamento. Possiamo idealmente suddividere il ribosoma in tre siti che svolgono differenti ruoli. Il sito P (peptidico) è quello dove si trova, temporaneamente, la catena in fase di allungamento; il sito A (aminoacidico) è dove viene condotto il nuovo aminoacido legato al tRNA da inserire nella catena; il sito E (exit) è il sito da dove viene espulso il tRNA.
Due fattori principali contribuiscono alla sintesi del polipeptide e sono EF-Tu ed EF-G. EF-Tu è legato ad un GTP ed ha il compito di legarsi all'aminoacil-tRNA formando un complesso ternario che viene condotto a livello del sito A del ribosoma; se il sito P del ribosoma è libero, ad esempio quando la sintesi sta iniziando, allora EF-Tu con la N-formilmetionina si legano al sito P. All'interno del ribosoma l'attività GTP-asica di EF-Tu idrolizza il GTP in GDP e come diretta conseguenza permette di lasciare il tRNA in sede e si dissocia dal ribosoma per ripristinare, al di fuori del ribosoma, il GTP.
Il secondo passo prevede la creazione del legame peptidico che è particolare in quanto coinvolge il gruppo carbossilico e aminico di due aminoacidi. Il legame peptidico è di tipo trans poiché l'ossigeno carbonilico e l'idrogeno si trovano in conformazione trans e, inoltre, ha una caratteristica risonanza che lo rende particolarmente stabile.
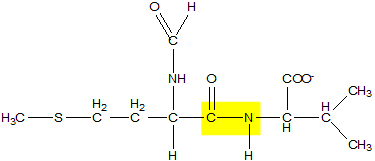
Legame peptidico tra N-formilmetionina e prolina
Quando il sito P è occupata il sistema EF-Tu-GTP-aminoacil-tRNA viene accettato dal ribosoma nel sito A; quando i due tRNA sono vicini avviene la reazione di sintesi del legame peptidico Questa reazione è catalizzata da un enzima strutturale del ribosoma chiamato peptidil transferasi che favorisce l'attacco dell'azoto al carbonio carbossilico.
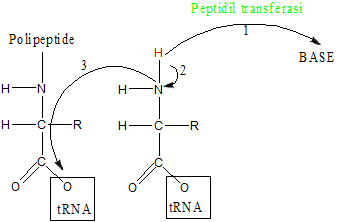
Probabile meccanismo di formazione del legame peptidico ad opera della peptidil transferasi
Il probabile meccanismo di formazione del legame peptidico prevede che la peptidil transferasi possa dapprima strappare un idrogeno al gruppo aminico e, di conseguenza, l'azoto fortemente reattivo andrebbe ad attaccare l'ossigeno ponte tra l'aminoacido della catena polipeptidica ed il tRNA.
Una volta che si formatosi il legame peptidico il ribosoma si sposta e la catena polipeptidica nascente viene traslocata sul sito A. Questo evento prende il nome di traslazione ed è fortemente velocizzato dal fattore EF-G che anch'esso è GTP dipendente.
A questo punto è opportuno fare alcune considerazioni. Negli eucarioti il complesso ribosomico riconosce il CAP che si trova all'estremità 5' ma nei procarioti tale struttura non è espressa; per questo motivo sarebbe plausibile un andamento di lettura dell'mRNA in direzione 3'→5'. In realtà in ambedue tipi cellulari la traduzione prosegue in direzione 5'→3' e, inoltre, il primo legame peptidico avviene tra il carbossile del primo aminoacido ed il gruppo aminico del secondo aminoacido per cui l'allungamento procede in direzione amino→carbossilica.
Terminazione
La terminazione avviene quando l'enzima incontra i codoni di stop che sono definiti dalle triplette UAA, UAG e UGA. A queste sequenze non si lega nessun tRNa ma speciali proteine le riconoscono ed ad esse si legano.
Nei procarioti tre fattori di rilascio, definiti RF1, RF2 ed RF3 mediano la localizzazione di uno dei codoni di stop e rendono possibile il rilascio del polipeptide. Negli eucarioti i fattori di rilascio sono due e sono definiti eRF1 ed eRF3.
|
Fattore
|
Ruolo
|
|
Procarioti
|
RF1
|
Riconosce e si lega ai codoni UUA ed UAG.
|
|
RF2
|
Riconosce e si lega ad UAA ed UGA.
|
|
RF3
|
Proteina GTP-dipendente che coadiuva il legame di RF1 o RF2 al ribosoma.
|
|
Eucarioti
|
eRF1
|
Riconosce i tre codoni di terminazione.
|
|
eRF3
|
Coadiuva il distacco del polipeptide dal ribosoma.
|
Fattori di rilascio procariotici ed eucariotici
Introduzione all'informazione genetica:
DNA e
RNA,
cromosoma,
cromatina,
genoma.
Trascrizione:
RNA polimerasi batterica,
allungamento dell'RNA,
Trascrizione eucariotica,
terminazione della trascrizione,
RNA interference (
pre-mRNA,
siRNA).
Traduzione:
Ribosoma,
RNA ribosomiale,
ribozima,
trascrittoma.
Duplicazione del DNA:
Primosoma,
forcella di replicazione,
DNA polimerasi I procariotica,
DNA polimerasi III procariotica,
Terminazione della duplicazione del DNA.
Tecniche e laboratorio PCR,
LCR,
Real time PCR.
Feedback
Il tuo aiuto è importante. Ti chiediamo un minuto per rispondere a questo breve sondaggio
Come valuteresti questo articolo?
Vuoi suggerirci qualcosa?