Lo splicing è un evento enzimatico nel quale vengono allontanati elementi di mRNA non codificante. L'mRNA non codificante è un tipo di mRNA che non ha alcuna valenza nel processo di sintesi proteica poiché non è tradotto al fine di generare sequenze di aminoacidi.
La molecola di mRNA immatura può contenere, nella propria sequenza, una linea di basi che non codificano per alcun aminoacido che vengono rimosse in alcuni eventi enzimatici.
Queste sequenze prendono il nome di sequenze non senso o introni. Gli esoni, a differenza, rappresentano le sequenze di basi che effettivamente possono codificare per un aminoacido.
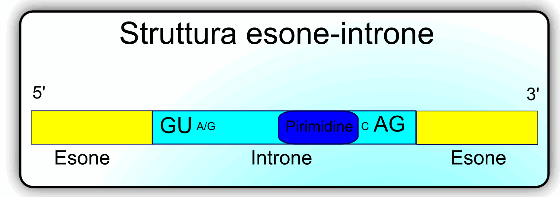
Struttura nucleotidica del sistema esone e introne. In testo grande i nucleotidi che sono soliti far parte dell'introne, in piccolo i nucleotidi la cui probabilità di presenza è alta.
Che cos'è lo splicing? È un processo coadiuvato dall'azione di specifici enzimi che hai il compito di far maturare l'mRNA allontanandone le sequenze non codificanti.
Quali sono le sequenze allontanate nello splicing? Tutte le sequenze tagliate via dallo splicing prendono il nome di introni.
Rimozione degli introni
Il processo di rimozione degli introni viene chiamato splicing e trova posto in specifici organuli chiamati spliceosomi anche se esistono delle particolarità in questi meccanismi. Grazie allo splicing, le sequenze non codificanti vengono allontanate dalla catena di RNA in una fase definita di maturazione. Alcuni geni sono del tutto assenti di sequenze che, una volta trascritte, formano zone di RNA intronico mentre altri geni possiedono più introni di lunghezza variabile.
Qual'è il segnale che determina la fine dell'esone e l'inizio dell'introne? Per determinare sperimentalmente queste sequenze nucleotidiche è stata di fondamentale aiuto la Statistica; una serie di comparazioni tra librerie di cDNA, acronimo di DNA complementare, e l'mRNA che hanno generato i frammenti di cDNA hanno permesso di determinare, con una ragionevole qualità, quali sono gli introni che rappresentano le sequenze non trascritte. Le analisi, inoltre, forniscono i valori di probabilità dei nucleotidi che determinano l'inizio degli introni e, in parte, la fine degli esoni. A questo proposito si è visto che per ciò che concerne l'introne questo è, quasi sempre, delimitato dalla sequenza:
5' GU---------AG 3'
All'interno dell'introne, a circa 30 basi di distanza della sequenza AG in 3', si trova una regione molto ricca in pirimidine.
Reazioni di splicing
Il meccanismo molecolare mediante il quale avviene lo splicing è ben conosciuto e coinvolge alcune transesterificazioni a livello della catena di pre-mRNA.
Dal punto di vista chimico le reazioni adoperate di tipo nucleofilo; in queste reazioni un atomo maggiormente elettronegativo, ad esempio un atomo di ossigeno, attacca un altro atomo meno elettronegativo, come ad esempio un carbonio. Le parti di mRNA allontanate non vengono utilizzate proprio perché non codificano per nessuna proteina e vengono degradate dalle nucleasi; in questo modo le preziose basi possono essere nuovamente riutilizzate.
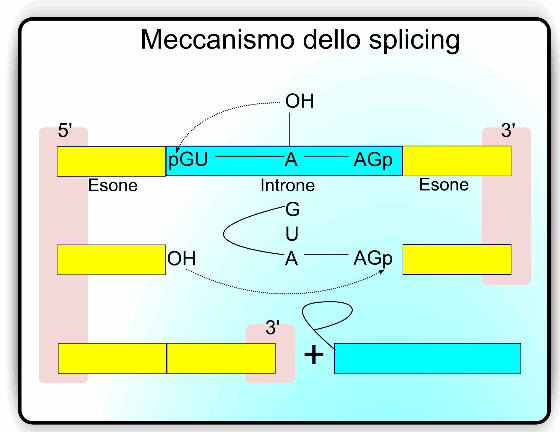
Reazioni e meccanismo d'azione dello splicing.
Una A, peculiare poiché si ritrova dentro l'introne, mediante il suo ossidrile in C-2, attacca il gruppo fosfato di giunzione tra la G dell'introne ed il resto dell'esone a monte. In questo modo si forma una catena corta di pre-RNA che si chiude su sé stessa, rimanendo legata all'esone a valle.
Una seconda trans-esterificazione tra l'ossidrile libero dell'esone - a monte - e il fosfato di giunzione dell'esone - a valle - che lega il lariat, che rappresenta la formazione a “laccio” precedentemente costituita, unisce i due esoni. A questo punto lo splicing è stato compiuto e l'estremità intronica, che mantiene la caratteristica struttura lariat, è allontanata.
Le reazioni appena viste non sono spontanee ma sono catalizzate da enzimi classificabili come ribonucleoproteine snRPS. Questi complessi, formati da RNA e da nucleoproteine formano gli spliceosomi, con differenti coefficienti di sedimentazione; nei mammiferi gli spliceosomi hanno un coefficiente pari a 60S.
Esistono cinque snRPS's denominate U1, U2, U4, U5 ed U6. U1 ed U2 si legano a monte dell'introne mentre U5 si lega a valle. U4 ed U6, invece, si legano assieme al complesso preformato. Lo spliceosoma ha la capacità di far avvicinare, dal punto di vista spaziale, le estremità notevoli del sistema esone-introne appena illustrato, per permettere gli attacchi nucloeofili e le conseguenti transesterificazioni a livello della catena di pre-mRNA da processare. Per compiere queste reazioni è richiesta energia sotto forma di ATP.
Introduzione all'informazione genetica:
DNA e
RNA,
cromosoma,
cromatina,
genoma.
Trascrizione:
RNA polimerasi batterica,
allungamento dell'RNA,
Trascrizione eucariotica,
terminazione della trascrizione,
RNA interference (
pre-mRNA,
siRNA).
Traduzione:
Ribosoma,
RNA ribosomiale,
ribozima,
trascrittoma.
Duplicazione del DNA:
Primosoma,
forcella di replicazione,
DNA polimerasi I procariotica,
DNA polimerasi III procariotica,
Terminazione della duplicazione del DNA.
Tecniche e laboratorio PCR,
LCR,
Real time PCR.
Feedback
Il tuo aiuto è importante. Ti chiediamo un minuto per rispondere a questo breve sondaggio
Come valuteresti questo articolo?
Vuoi suggerirci qualcosa?